Kekhususan deteksi
Umume kasus, tujuan desain primer yaiku kanggo nggedhekake kekhususan PCR.Iki ditemtokake dening pengaruh luwih utawa kurang bisa diprediksi saka akeh variabel.Salah sawijining variabel penting yaiku urutan ing 3′ pungkasan primer.
Sing penting, tes PCR sing dirancang kanggo spesifisitas luwih cenderung njaga efisiensi sing dhuwur sajrone rentang dinamis sing amba, amarga tes kasebut ora ngasilake produk amplifikasi sing ora spesifik, mula saingan karo reagen PCR utawa nyegah reaksi amplifikasi utama.
Mesthi, ing sawetara kasus, spesifisitas ora sing paling penting, contone, nalika tujuane kanggo ngitung sing gegandhengan nanging patogen beda, desain khusus, optimasi lan standar verifikasi dibutuhake.
Kurva leleh minangka cara standar kanggo ngevaluasi kekhususan amplikon, paling ora babagan nggedhekake target siji.Nanging, iku kudu nandheske sing kurva leleh bisa mblusukake amarga, contone, padha bisa kena pengaruh saka efek gabungan saka primer suboptimal lan konsentrasi Cithakan kurang.
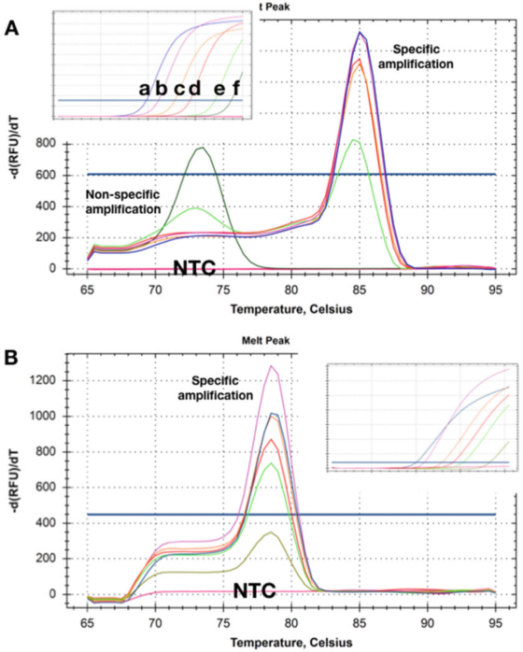
P5 |Kurva leleh nuduhake owah-owahan Tm sing dipikolehi saka rong deteksi jumlah sing beda saka rong DNA target.
A. Ing konsentrasi sing luwih dhuwur (ad)), ora ana dimer primer sing jelas sawise pangukuran qPCR rampung.Nalika konsentrasi Cithakan sudo kanggo 50 salinan (e), produk non-tartamtu wiwit katon lan dadi mung produk ing konsentrasi paling (f).
B. Tes kasebut nyathet Tms sing padha ing kabeh konsentrasi target, lan ora ana dimer primer sing jelas sanajan ing konsentrasi paling murah (5 salinan).Nalika nggunakake rong cara deteksi iki, ora ana produk amplifikasi sing dideteksi ing NTC.
P5 nuduhake kurva pembubaran sing dipikolehi karo conto ing ngendi cithakan kasebut ana ing konsentrasi sing beda.P 5a nuduhake yen ing rong konsentrasi paling murah, Tms produk amplifikasi non-spesifik sing diasilake luwih murah tinimbang amplikon spesifik.
Temenan, cara deteksi iki ora bisa digunakake kanthi dipercaya kanggo ndeteksi target sing ana ing konsentrasi sing sithik.
Sing nggumunake, NTC, yaiku, conto sing ora ana DNA, ora ngrekam produk amplifikasi (non-spesifik), sing nuduhake yen DNA genom latar mburi bisa melu amplifikasi/polimerisasi non-spesifik.
Kadhangkala primer latar mburi lan amplifikasi non-tartamtu kuwi ora bisa remedied, nanging asring bisa kanggo desain cara deteksi sing ora duwe amplifikasi non-tartamtu ing sembarang konsentrasi cithakan lan NTC (P 5b).
Ing kene, sanajan ngrekam amplifikasi konsentrasi target kanthi Cq 35 bakal ngasilake kurva pembubaran tartamtu.Kajaba iku, NTC ora nuduhake tandha-tandha amplifikasi non-spesifik.Kadhangkala, prilaku deteksi bisa uga gumantung marang omben-omben ibu, lan mung amplifikasi non-spesifik sing dideteksi ing komposisi buffer tartamtu, sing bisa uga ana hubungane karo konsentrasi Mg2 + sing beda.
Stabilitas deteksi
Optimisasi Ta minangka langkah migunani ing verifikasi empiris lan proses optimasi deteksi qPCR.Nyedhiyakake indikasi langsung saka kekokohan set primer kanthi nuduhake suhu (utawa sawetara suhu) sing ngasilake Cq paling murah tanpa nggedhekake NTC.
Bedane sensitivitas loro nganti patang lipat bisa uga ora penting kanggo wong kanthi ekspresi mRNA sing dhuwur, nanging kanggo tes diagnostik, bisa uga tegese bedane asil negatif positif lan palsu.
Sifat Ta saka primer qPCR bisa beda-beda banget.Sawetara tes ora kuwat banget, lan yen ora dileksanakake miturut nilai Ta optimal saka primer, bakal cepet ambruk.
Iki penting amarga deteksi jinis iki asring dadi masalah ing jagad nyata, lan kemurnian sampel, konsentrasi DNA, utawa anané DNA liya bisa uga ora optimal.
Kajaba iku, nomer salinan target bisa beda-beda ing macem-macem, lan reagen, piranti plastik, utawa instrumen bisa uga beda karo sing digunakake nalika nyetel tes.
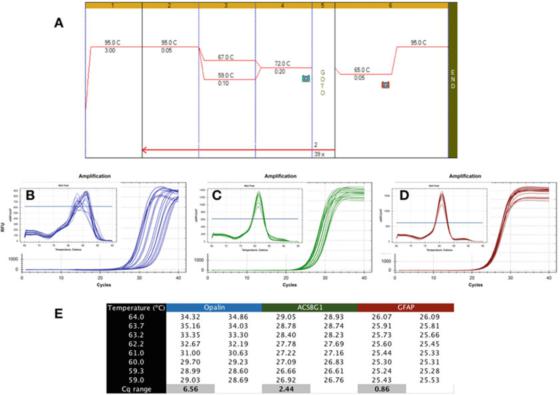
P6|Gredean suhu nuduhake kakuwatan deteksi PCR sing beda.
A. Gunakake mastermix Sensifast SYBR Bioline (nomer katalog BIO-98050) kanggo nindakake PCR ing cDNA sing disiapake saka RNA otak manungsa.
B. Gunakake instrumen CFX qPCR Bio-Rad kanggo ngrekam peta amplifikasi lan kurva pembubaran apalene (NM_033207, F: GCCATGGGAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGATCTCCTAGG).
C. Grafik amplifikasi lan kurva leleh saka ACSBG1 (NM_015162.4, F: CTACACTTCCGGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. Grafik amplifikasi lan kurva pembubaran GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs direkam ing suhu annealing beda, nuduhake prabédan ing Cq direkam ing gradien suhu 7C.
P 6 nuduhake asil khas saka tes sing ora dikarepake, ing ngendi qPCR ditindakake kanthi nggunakake gradient Tas antarane 59C lan 67C (P 6a), nggunakake primer kanggo telung gen spesifik otak manungsa.
Bisa dideleng saka grafik amplifikasi yen primer Opalin adoh saka ideal amarga rentang Ta optimal banget sempit (Gambar 6b), yaiku, Cqs disebarake kanthi akeh, nyebabake Cqs dibandhingake karo Cqs Low optimal.
Cara deteksi iki ora stabil lan bisa nyebabake amplifikasi suboptimal.Mulane, pasangan primer iki kudu didesain ulang.Kajaba iku, analisis kurva leleh (inset) nuduhake yen kekhususan metode deteksi iki uga bisa dadi masalah, amarga kurva leleh saben Ta beda-beda.
Cara deteksi ACSBG1 sing ditampilake ing P 6c luwih kuat tinimbang metode deteksi Opalin ing ndhuwur, nanging isih adoh saka ideal, lan kemungkinan bisa ditingkatake.
Nanging, kita nandheske yen ora ana sambungan sing perlu antarane kekokohan lan kekhususan, amarga kurva pembubaran sing diprodhuksi dening metode deteksi iki nuduhake nilai puncak sing padha ing kabeh Tas (inset).
Ing sisih liya, tes kekiatan luwih toleran, ngasilake Cq sing padha ing macem-macem Tas, kaya ing tes GFAP sing ditampilake ing P 6d.
Bentenipun ing Cqs dijupuk ing padha 8 derajat Celsius sawetara kurang saka 1, lan kurva disolusi (inset) nandheske karakteristik deteksi ing sawetara suhu iki.Wigati dicathet menawa Tas sing diwilang lan sawetara Ta nyata bisa uga beda banget.
Ana akeh pedoman sing dirancang kanggo mbantu peneliti ngrancang primer sing efisien, sing paling akeh adhedhasar aturan sing wis suwe lan akeh perhatian wis dibayar kanggo 3′ pungkasan primer.Asring dianjurake kanggo kalebu G utawa C ing mburi 3 'lan loro G utawa C basa (GC clamp), nanging ora luwih saka loro saka 5 basa pungkasan.
Ing laku, aturan kasebut bisa nuntun peneliti, nanging ora mesthi bener ing kabeh kahanan.
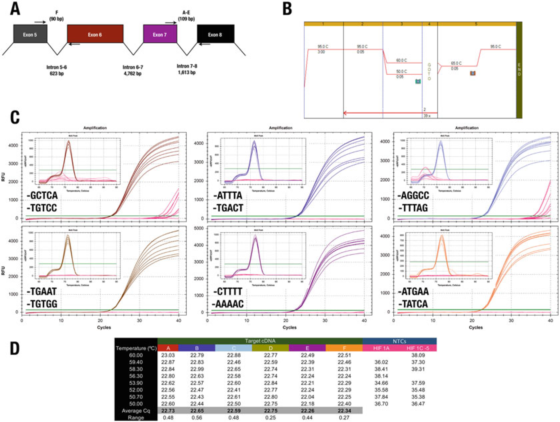
P7 |Pungkasan 3′ sepisanan nduweni pengaruh cilik ing spesifik utawa efisiensi.
A. Posisi primer kanggo gen HIF-1α (NM_181054.2) manungsa.
B. Gunakake Agilent Brilliant III SYBR Green omben-omben ibu (Cat. No. 600882) kanggo nggedhekake enem item test.
C. Grafik amplifikasi lan kurva leleh sing direkam dening instrumen CFX qPCR Bio-Rad lan primer 3′end.NTC ditampilake kanthi warna abang.
D. Cqs cathetan saben item test
Contone, asil ing P 7 mbantah aturan 3′end.Kabeh desain ngasilake asil sing padha, kanthi mung rong kombinasi primer sing ndadékaké amplifikasi non-spesifik ing NTC.
Nanging, kita ora bisa ndhukung efek saka klip GC, amarga ing kasus iki, nggunakake A utawa T minangka maksimum 30 basa ora nyuda specificity.
Test C, ngendi sepisanan F ends ing GGCC, nyathet Cqs ing NTCs, nuduhake yen siji bisa uga pengin supaya urutan iki ing 30-mburi.We nandheske sing siji-sijine cara kanggo nemtokake urutan paling apik 3′end saka pasangan primer iku kanggo ngira-ngira sawetara primers calon eksperimen.
Efisiensi amplifikasi
Sing penting, sanajan deteksi PCR sing ora spesifik ora bisa dadi spesifik, efisiensi amplifikasi bisa diatur lan dimaksimalake kanthi macem-macem cara kanthi ngganti enzim, omben-omben ibu, aditif, lan kondisi siklus.
Kanggo ngevaluasi efisiensi deteksi PCR, luwih becik nggunakake pengenceran serial 10 utawa 5 kaping asam nukleat target, yaiku, "metode kurva standar".
Yen amplikon PCR utawa target DNA sintetik digunakake kanggo ngasilake kurva standar, pengenceran serial target kasebut kudu dicampur karo jumlah DNA latar mburi sing konstan (kayata DNA genom).
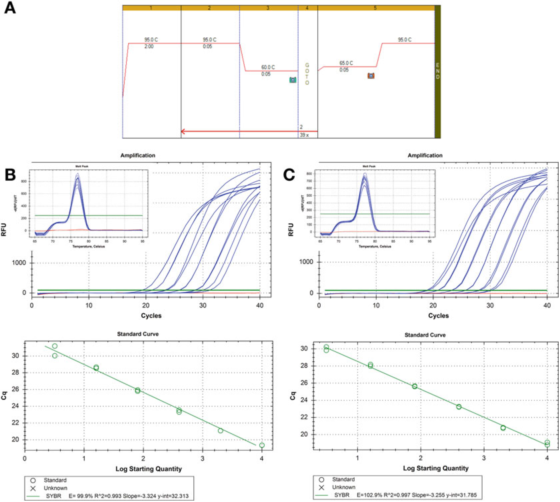
P8 |Kurva pengenceran kanggo ngevaluasi efisiensi PCR.
A. Gunakake primer kanggo HIF-1: F: AAGAACTTTTAGGCCGCTCA lan R: TGTCCTGTGGTGACTTGTCC lan mastermix Agilent kang Brilliant III SYBR Green (nomer katalog 600882) kanggo PCR lan kondisi kurva leleh.
B. 100 ng RNA iki mbalikke ditranskripsi, diencerke 2 kaping, lan serially diencerke cDNA conto diencerke 5 kaping kanggo 1 ng DNA genomik manungsa.Kurva leleh ditampilake ing inset.
C. Reaksi RT, pengenceran, lan pengenceran serial diulang kanggo sampel cDNA kapindho, lan asile padha.
P 8 nuduhake loro kurva standar, nggunakake cara deteksi padha ing loro conto cDNA beda, asil efficiency padha, bab 100%, lan Nilai R2 uga padha, sing, jurusan Pas antarane data eksperimen lan baris kemunduran utawa data Degree of linearity.
Loro kurva standar bisa dibandhingake, nanging ora persis padha.Yen tujuane kanggo ngitung target kanthi akurat, kudu dicathet yen ora bisa ditampa kanggo menehi pitungan nomer salinan tanpa nerangake kahanan sing durung mesthi.
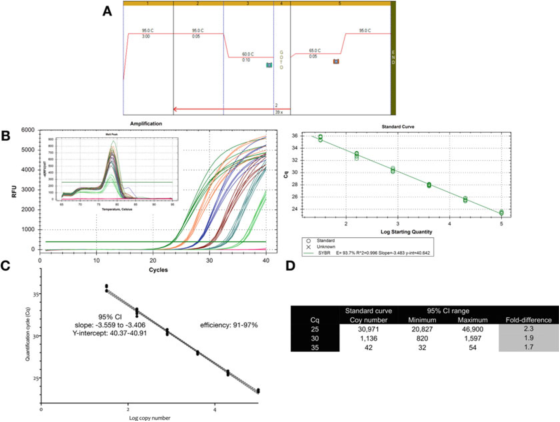
P9 |Ketidakpastian pangukuran sing ana gandhengane karo kuantifikasi nggunakake kurva standar.
A. Gunakake primer kanggo GAPDH (NM_002046) kanggo nindakake PCR lan kondisi kurva leleh.F: ACAGTTGCCATGTAGACC lan R: TAACTGGTTGAGCACAGG lan mastermix Sensifast SYBR Bioline (nomer katalog BIO-98050).
B. Bagan amplifikasi, kurva leleh lan kurva standar sing direkam nganggo instrumen qPCR CFX Bio-Rad.
C. Grafik kurva standar lan interval kapercayan 95% (CI).
D. Nomer salinan lan interval kapercayan 95% saka telung nilai Cq sing diturunake saka kurva pengenceran.
P 9 nuduhake yen kanggo tes sing dioptimalake, variasi inheren saka kurva standar siji kira-kira 2 kali (interval kapercayan 95%, minimal nganti maksimal), sing bisa dadi variabilitas paling cilik sing bisa diarepake.
produk sing gegandhengan:
Wektu kirim: Sep-30-2021